ggrepel-解决散点图样品标签重叠,方便筛选样品
共 3172字,需浏览 7分钟
· 2022-07-04
ggrepel解决标签之间重叠问题
简介
有时样本比较多,而我们想在图形中添加标签的时候,容易出现标签遮盖的问题。
尤其是在扩增子研究中,在相同基因型、环境条件宿主(温室植物、饲养动物)至少也需要6次以上生物学重复,如人类这种无法控制基因型和生活环境的研究对象,实验组至少30个起才容易发现有统计为意义的差异菌。
而在样品比较、样品筛选时又必须看清这些点名字,用于筛选掉一些记录错误、未报抗生素使用或隐性疾病等异常样品。ggplot2的辅助包ggrepel就是专门处理遮盖问题的专家。有了人类可读的可视化结果,在我们下游分析、样品筛选、异常样品鉴定更加方便高效。
ggrepel(https://github.com/slowkow/ggrepel)是发表在github上的开源包,使用之前是要先安装:
安装
Rstudio中安装稳定版本:
install.packages("ggrepel")
# 如果在R中,需要选择源或指定源
install.packages("ggrepel", repo="http://cran.us.r-project.org")或者安装最新的开发版本:
install.packages("devtools", repo="http://cran.us.r-project.org")
library(devtools)
devtools::install_github("slowkow/ggrepel")geom_text()添加样品标签
我们先看看geom_text()添加标签时的效果
library(ggplot2)
#使用系统数据集mtcars演示
ggplot(mtcars)+ geom_point(aes(wt, mpg), color="red")+
geom_text(aes(wt, mpg, label=rownames(mtcars)))+
theme_classic(base_size = 16)
可以看到可视化效果不是很好。接下来看看包ggrepel的效果。
geom_text_repel()解决样品标签重叠
geom_text_repel()是基于geom_text()
library(ggrepel)
set.seed(123)
ggplot(mtcars)+ geom_point(aes(wt, mpg), color="red")+
geom_text_repel(aes(wt, mpg, label=rownames(mtcars)))+
theme_classic(base_size = 16)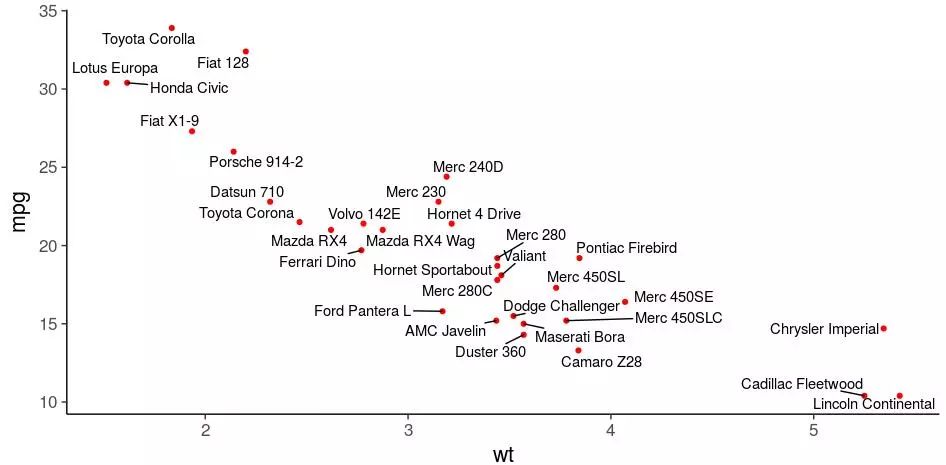
geom_label_repel()防标签重叠并添加背景色
geom_label_repel()是基于geom_label(),它将标签置于一个小方框中
ggplot(mtcars)+ geom_point(aes(wt, mpg), color="grey", size=5)+
geom_label_repel(aes(wt, mpg, fill=factor(cyl),
label=rownames(mtcars)))+ theme_classic(base_size = 16)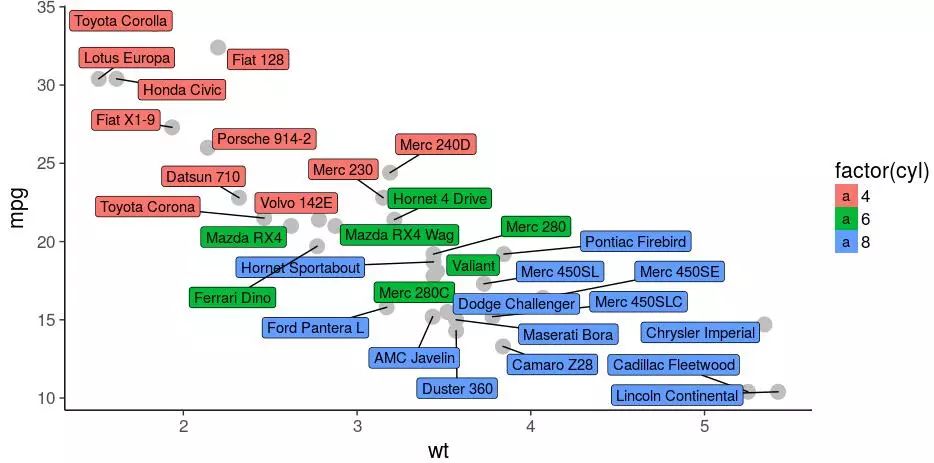
点太小颜色不容易区分组,直接给标签上色是不是很容易区分样品和组,以及观察组内和组间的差异、筛选异常样品呢?
基于扩增子分析PCoA实战数据
测试数据和代码详见下文:
我们在此基础上添加标签、错开标签,以及按标签着色筛选样品。
geom_text添加样品名
# 绘制主坐标准轴的第1,2轴
p = ggplot(points, aes(x=x, y=y, color=genotype)) +
geom_point(alpha=.7, size=2) +
labs(x=paste("PCoA 1 (", format(100 * eig[1] / sum(eig), digits=4), "%)", sep=""),
y=paste("PCoA 2 (", format(100 * eig[2] / sum(eig), digits=4), "%)", sep=""),
title="bray_curtis PCoA")
p + geom_text(aes(x, y, label=rownames(points)))+ theme_classic()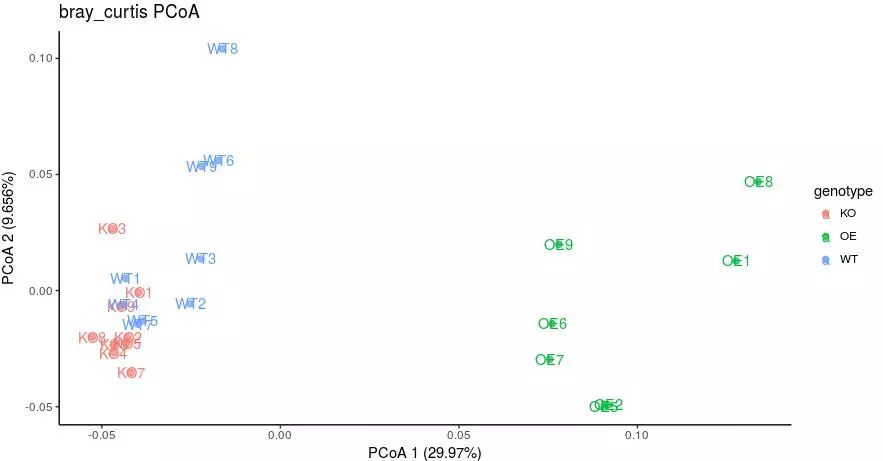
够乱吧,根本看不清。
geom_text_repel合理位置添加样品名
library(ggrepel)
p + geom_text_repel(aes(x, y, label=rownames(points)))+ theme_classic()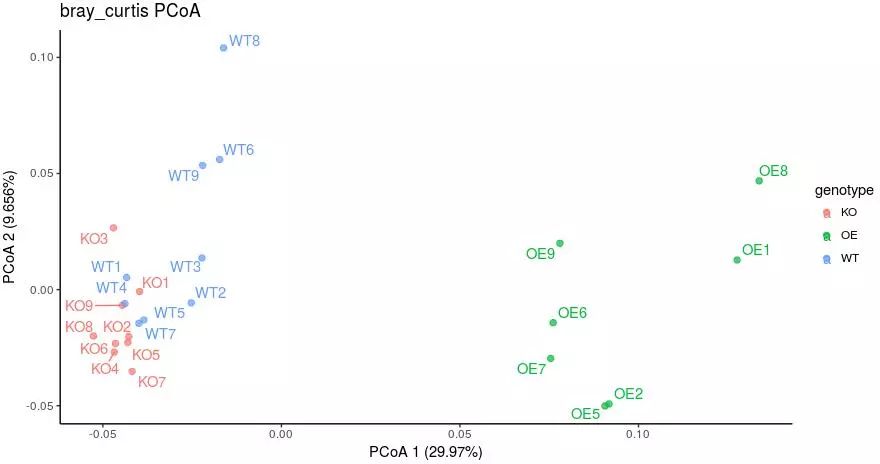
好多了吧!
geom_label_repel合理位置添加标签
需要调整文字和点不上色,只按标签背景填充色,代码如下:
ggplot(points, aes(x=x, y=y)) +geom_point(alpha=.7, size=2) + geom_label_repel(aes(x, y, fill=factor(genotype), label=rownames(points)))+ theme_classic()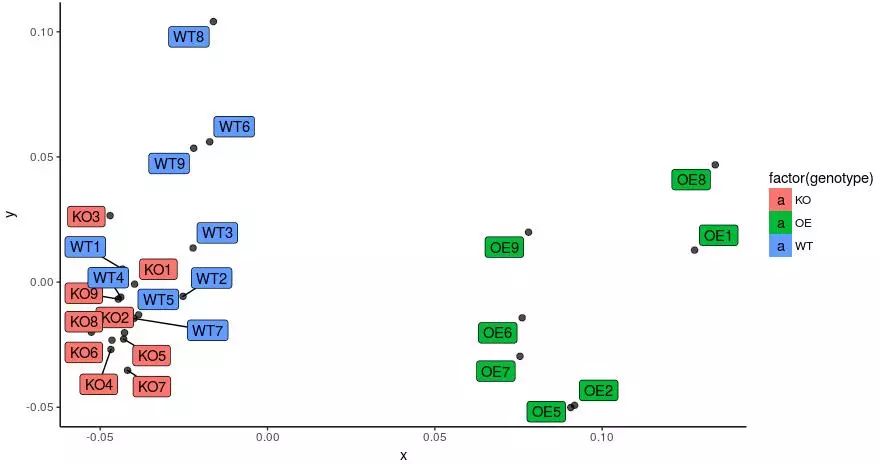
另一种上色方式,按标签背景分组上色,好像选择样品看容易,比点着色看的清楚。
现在可以一眼看到异常样品的位置了。如果还无法确定,可以结合PCA和hculst的聚类结果综合排除异常样品。想在此图中对分组进一步添加置信区间,方便显示组间是否有差异,以及定义圈外异常样品,将在过几天与大家分享。
Reference
官方包下载和教程 https://github.com/slowkow/ggrepel
孙老湿画图系列第十一弹丨标签遮盖处理工具ggrepel http://baijiahao.baidu.com/s?id=1576516080050548076&wfr=spider&for=pc
R语言可视化学习笔记之ggrepel包 https://mp.weixin.qq.com/s/ZKxzKZ4NBTcsJ6vFimxoGA?scene=25#wechat_redirect
推荐阅读
学习津贴
单篇留言点赞数的第一位(点赞数至少为8)可获得我们赠送的在线基础课的9折优惠券。
越留言,越幸运。
主编会在每周选择一位最有深度的留言,评论者可获得我们赠送的任意一门在线课程的9折优惠券(偷偷告诉你,这个任意是由你选择哦)。
高颜值免费在线绘图
往期精品
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集
